Kontext - Pilzerkennung
Pilzerkennung: Eine der am weitesten verbreiteten und ältesten Artenbestimmungsmethoden ist die „morphologische Bestimmung“, bei der die Individuen anhand ihrer anatomischen Merkmale identifiziert werden. Diese Technik hat jedoch den Nachteil, dass sie nicht immer genau ist, da sie von der Beobachtung und dem Bestimmungsprotokoll der Person abhängt, die sie durchführt.
Eine Alternative zu dieser Identifizierungstechnik könnten Computer-Vision-Algorithmen sein. Diese Algorithmen ermöglichen es uns, die Merkmale eines Bildes schnell und genau zu erkennen, so dass die Identifizierung einer Art nur durch ein Foto erfolgen kann, was sie leichter übertragbar macht.
Im Folgenden versuchen wir, verschiedene Pilzbilder mit Hilfe von Computer Vision zu identifizieren. Diese Arbeit wurde zusammen mit meinem Promotionskollegen Cyril Vandenberghe durchgeführt und von Adrien Ruimy betreut. Sie entspricht dem Abschlussprojekt des DataScientest-Kurses für Datenwissenschaftler. Die Entwicklung dauerte hundert Stunden und ermöglichte es uns, unser neu erworbenes Wissen in einer praktischen Anwendung zu nutzen.
Projektautoren: Karina Castillo & Cyril Vandenberghe.
Das Projekt
Data source
Für dieses Projekt haben wir Daten von mushroomobserver.org verwendet, einer Website, die Bilder von Pilzen sammelt und identifiziert: Die Nutzer laden Bilder hoch, und die Community identifiziert sie. Derzeit zählt die Website über 10.000 registrierte Nutzer.
Bei unserer anfänglichen Literaturrecherche stellten wir fest, dass ein früheres Projekt auf Github, “ mushroomobser-dataset“, diese Seite bereits erkundet hatte, um einen Datensatz mit Bildern aus den Jahren 2006 bis 2016 zu erstellen. Außerdem enthalten 12 JSON-Dateien zusätzliche Informationen zu jedem Bild.

Data Exploration
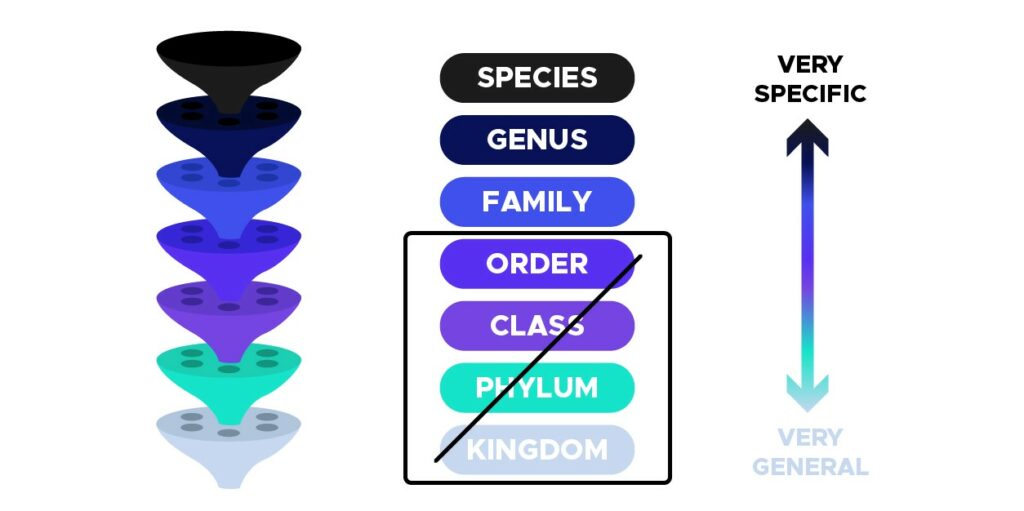
Der erste Schritt bei der Verarbeitung unserer Daten bestand darin, einen Datenrahmen mit Informationen über die Bilder und die Taxonomie der Pilze zu erstellen. In der Biologie ist eine Taxonomie eine Möglichkeit, hierarchische Gruppen auf der Grundlage gemeinsamer Merkmale zu definieren, wie in der Abbildung unten dargestellt:

In der Tabelle 1 zeigt die Spalte „Rang“ die letzte taxonomische Ebene an, die bei der Pilzbestimmung in unserem Datenrahmen erreicht wurde. Wenn also ein Eintrag in der Spalte `Rang` den Wert `KINGDOM` hat, ist der Wert in der Spalte `Label` die allgemeinste Identifikation, z.B. `Fungi`. Die identifizierten Bilder können also zu jedem Pilz gehören.
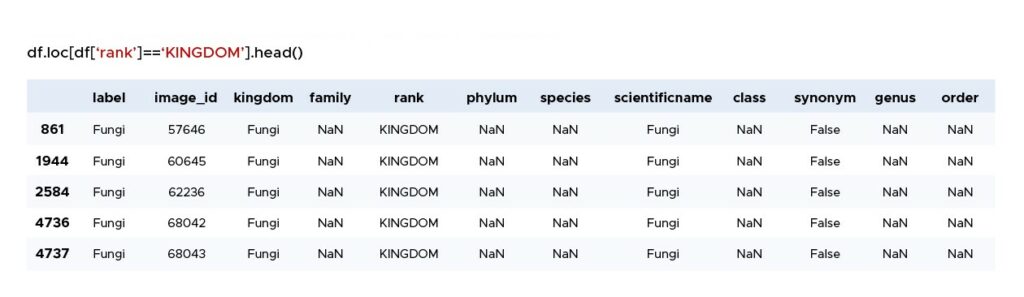
Wenn die Spalte `Rang` dagegen `SPECIES` (ein spezifischeres Taxon) ist, haben wir eine detailliertere Beschreibung der Bilder (Tabelle 2).
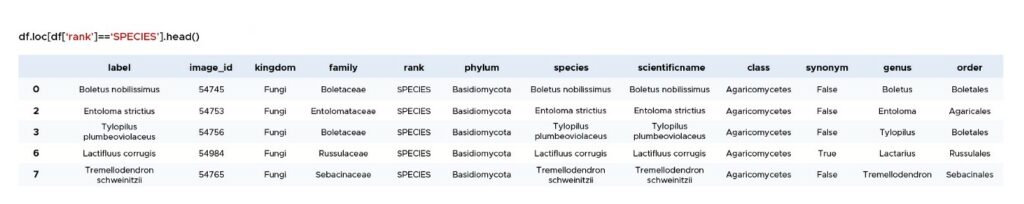
Daher wurden Bilder mit den Werten `KINGDOM`, `PHYLUM`, `CLASS` und `ORDER` in der Spalte `rank` verworfen, da sie zu allgemein und nicht informativ sind. Im weiteren Verlauf des Projekts werden wir nur noch mit Daten arbeiten, die zu den Taxa SPECIES, GENUS und FAMILY gehören, wie in der folgenden Abbildung dargestellt:
Data Visualisation
Eine große Anzahl von Bildern ist für die erfolgreiche Entwicklung eines Computer Vision Projekts unerlässlich. In diesem Projekt benötigen wir Labels (d.h. Werte in der Spalte „Label“), um eine große Anzahl von Bildern für jedes Taxon zu erhalten.
Die folgende Abbildung (Abb. 4) zeigt die Verteilung der Anzahl der Bilder für die Taxa. Jedes der vier Felder zeigt die Anzahl der Bezeichnungen (y-Achse) für ein bestimmtes Taxon (x-Achse), dargestellt durch 1 bis 10 Bilder, 11 bis 100 Bilder, 101 bis 1000 Bilder und über 1000 Bilder.
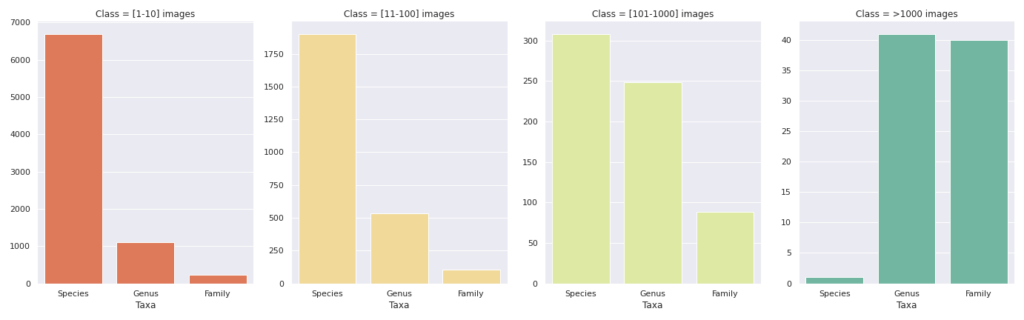
Das Artentaxon ist das ideale, weil es das spezifischste ist. Allerdings entspricht die Anzahl der Labels, die nur durch 1 bis 10 Bilder repräsentiert werden, 75 % der Gesamtzahl der Arten. Im Intervall [101-1000] haben nur sieben Arten mehr als 500 Bilder, und nur eine Art hat mehr als 1000 Bilder.
Im Panel für Kategorien mit mehr als 1000 Bildern beobachten wir dagegen die Taxa Gattung und Familie mit jeweils etwa 40 Bezeichnungen, was sie zu guten Zielen macht. Taxonomisch gesehen ist die Familie die am wenigsten spezifische Ebene, so dass wir eine größere Heterogenität der Bilder erwarten würden, wenn wir diese Ebene wählen. Die Gattung scheint also ein guter Kompromiss zwischen der taxonomischen Ebene und der Anzahl der verfügbaren Bilder zu sein.
Final Dataset : Genus Taxa
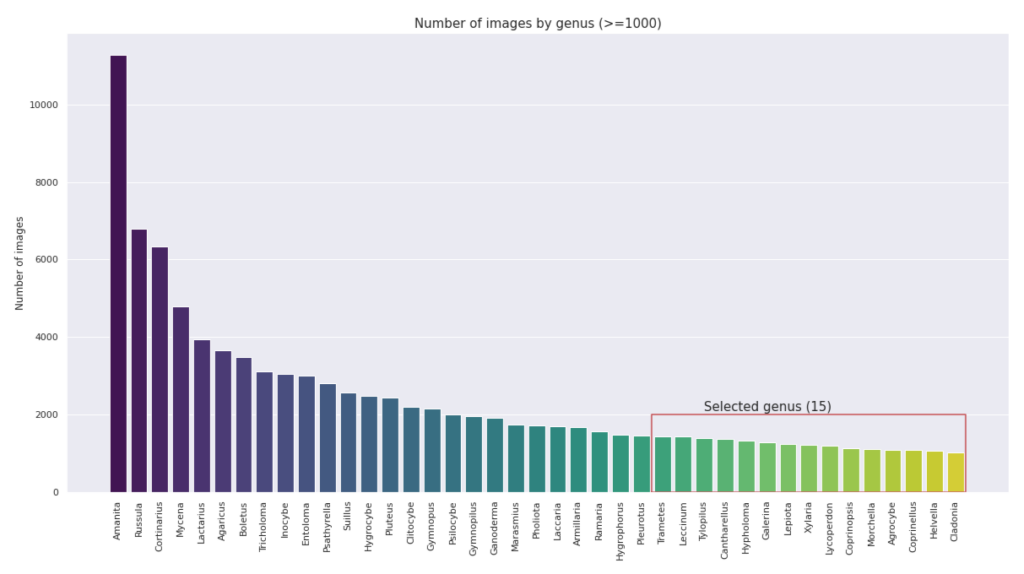
Das Balkendiagramm unten zeigt die Anzahl der Bilder für 41 ausgewählte Gattungen (Abb. 5). Wir haben beschlossen, uns auf die 15 hervorgehobenen Gattungen mit ~1.000 Bildern zu beschränken, um die Berechnungszeit zu reduzieren und eine verzerrte Darstellung zu vermeiden. Damit haben wir insgesamt 18.412 Bilder für den Klassifizierungs-/Modellierungsteil unseres Projekts zur Verfügung.
Klassifizierung mit neuronalen Netzen
Architekturen
Wie bereits erwähnt, zielt dieses Projekt darauf ab, Pilzbilder zu identifizieren. Das ist ein typisches Bildklassifizierungsproblem, das in der Regel mit Deep-Learning-Methoden angegangen wird. Um unser Ziel zu erreichen, haben wir drei Modelle getestet, die im Folgenden beschrieben werden.
LeNet
Zunächst haben wir unsere Daten mit einem CNN-Modell mit LeNet-Architektur (Y. LeCun et al., 1998) getestet, das für die Bildverarbeitung angepasste Schichten enthält. Wir haben dieses Modell mit zwei Faltungsschichten getestet.
Mit einem Wert von 0,98% Genauigkeit scheint das Basismodell für unser Ziel nicht geeignet zu sein, daher haben wir dieses Modell nicht weiter verfolgt.
VGG16
Als Nächstes verwendeten wir das VGG16-Modell (Simonyan und Zisserman, 2015), ein Bildklassifizierungsmodell mit einem Transfer Learning-Ansatz.
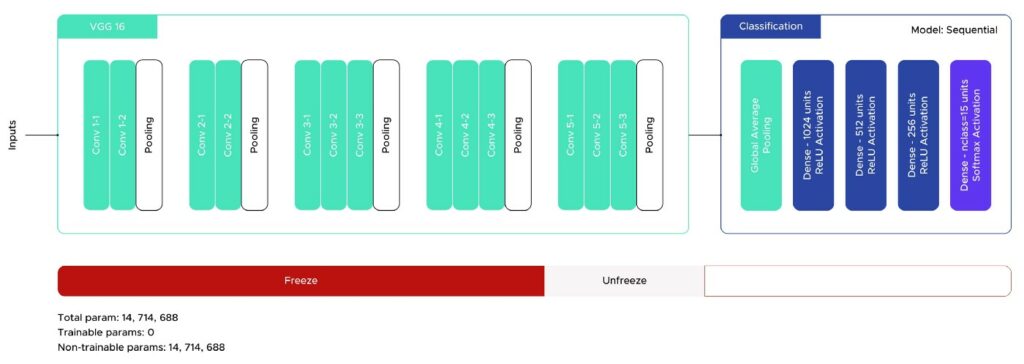
Im ersten Schritt hatte unser Modell eine Klassifizierungsschicht mit einem sequentiellen Modell, zwei neuronale Schichten (Dense) und zwei Regularisierungsschichten (Dropout).
Die erzielten Ergebnisse waren ermutigend, mit einer Genauigkeit von etwa 70 %, aber das Modell litt unter einer Überanpassung. Um dem entgegenzuwirken, fügten wir eine Dense-Schicht hinzu und erhöhten den Dropout-Wert. Abbildung 6 zeigt die endgültige Architektur des Modells.
Zusätzlich haben wir die Callbacks EarlyStopping und ReduceLROnPlateau aus dem Keras-Paket mit Geduldswerten von 5 bzw. 3 verwendet. Damit erreichten wir eine Genauigkeit von 75,5 % ohne Overfitting.
EfficientNetB1
Die dritte Methode zur Bildklassifizierung, die wir in diesem Projekt verwendeten, war ebenfalls ein Transfer Learning-Ansatz mit dem Tensorflow-Modell EfficientNet (Tan und Le, 2019). Bei der implementierten Klassifizierungsarchitektur haben wir uns von unserem vorherigen Modell (VGG16) inspirieren lassen, da wir damit gute Ergebnisse erzielt haben (Abb. 7). Wir haben auch die Callbacks EarlyStopping und ReduceLROnPlateau mit denselben Werten verwendet.
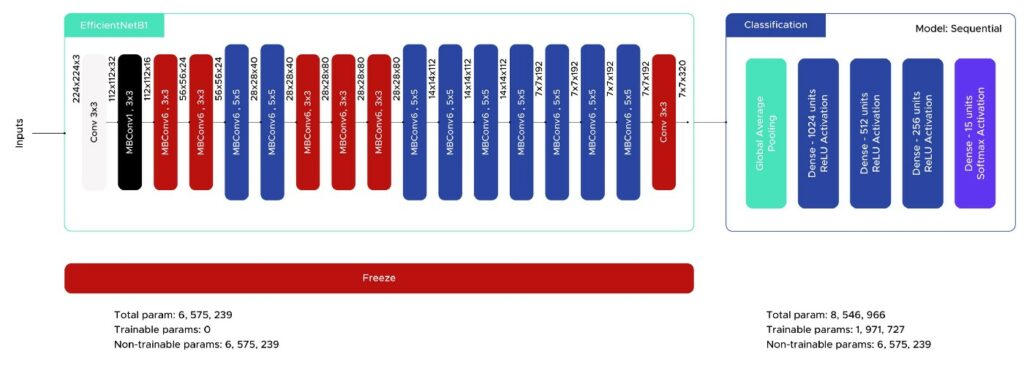
Die ersten Ergebnisse zeigen eine Genauigkeit von etwa 80 %, besser als bei VGG16, aber immer noch eine Überanpassung. Deshalb haben wir den Wert der Dropout-Schichten erhöht und schließlich ein Ergebnis von 79 % ohne Überanpassung erzielt.
Hier siehst du die Vorhersagen des Modells für sechs Bilder:

Error Analysis
Konfusionsmatrix
Die Konfusionsmatrix (Abb. 9) zeigt, dass die Klassen 0, 3 und 8 eher den Klassen 7, 4 bzw. 13 zugeordnet werden:
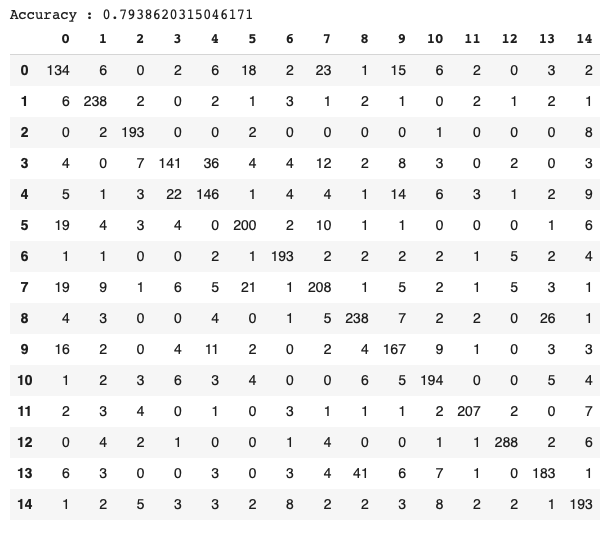
Die Literatur zeigt, dass diese verwechselten Klassen morphologisch sehr ähnlich sind und zur selben taxonomischen Familie gehören (Tabelle 3).
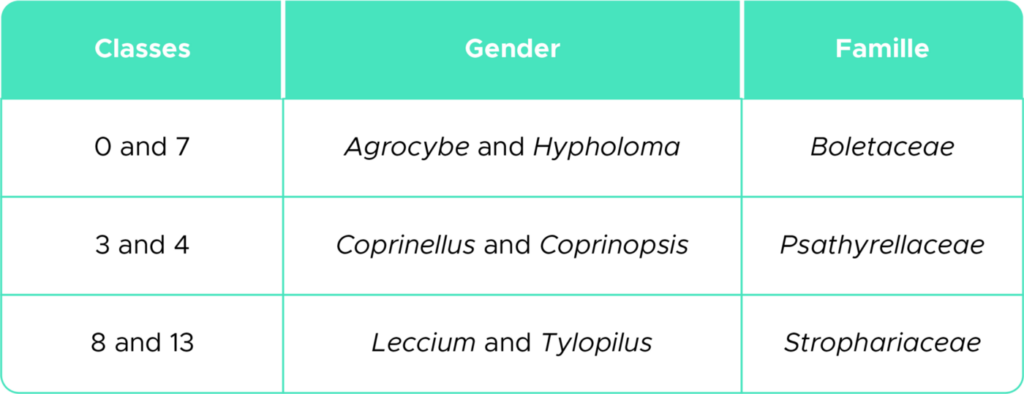
Die Abbildung 10 zeigt diese morphologischen Ähnlichkeiten zwischen den verwechselten Klassen:

Ein Grund für die Verwirrung der Klassen kann sein, dass die taxonomische Identifizierung der Pilze falsch ist. Die Bilder, die wir verwenden, stammen von einer Webseite, auf der die Nutzer Bilder von Pilzen nur durch das Betrachten der Bilder und nach ihrer Erfahrung klassifizieren und nicht nach einem objektiven taxonomischen Bestimmungsprotokoll.
Angesichts der Ergebnisse der Konfusionsmatrix wollten wir unser Modell ohne eine der verwechselten Gattungen bewerten, um den Einfluss der Daten auf unser Modell zu sehen (siehe Tabelle 4).

Der Genauigkeitswert für die 12 Gattungen (ohne die drei ausgeschiedenen) liegt bei 85 %, 6 % höher als das vorherige Ergebnis (79 %). Dieses Ergebnis deutet darauf hin, dass wir unsere Genauigkeit wahrscheinlich erhöhen würden, wenn wir einen genaueren Datensatz von Klassifizierungen zum Trainieren des Modells gehabt hätten.
Qualität der Bilder
Wir haben auch festgestellt, dass die Qualität der Bilder sehr unterschiedlich ist (Abb. 11). Einige Bilder enthalten Elemente, die das Modell daran hindern können, den Pilz richtig zu identifizieren, z. B. Bilder mit viel Rauschen um den zu klassifizierenden Pilz (Bilder 1 und 2). Bilder, auf denen kein Pilz zu erkennen ist (Bild 3). Bilder mit mehr als einem Pilz (Bild 4).

Fazit
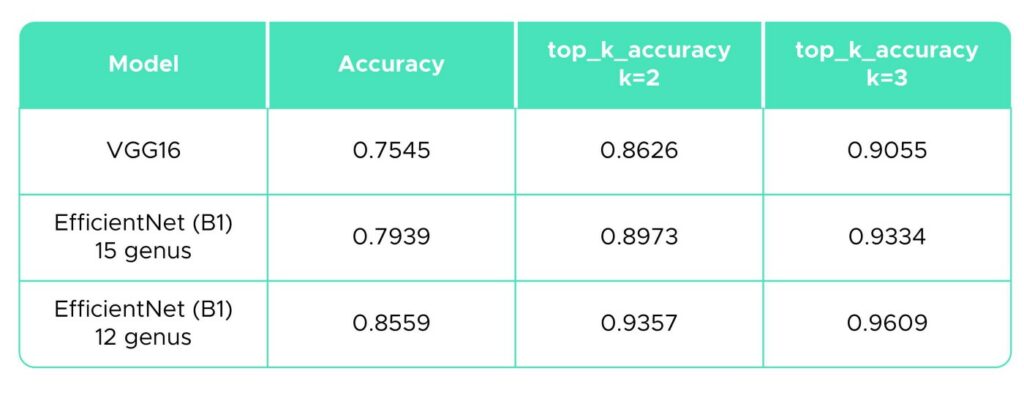
Die folgende Übersichtstabelle (Tabelle 5) zeigt die Genauigkeitswerte für die drei verwendeten Methoden. Wir haben auch den „top-k accuracy classification score“ des Pakets sklearn.metrics (Pedregosa et al., 2011.) mit k = 2 und 3 berechnet, um zu beurteilen, wie oft die tatsächliche Kategorie zwischen den zwei oder drei wahrscheinlichsten Kategorien liegt. Den Ergebnissen zufolge wäre EfficientNet mit 80 % und 86 % Genauigkeit der am besten geeignete Ansatz für unser Pilzbild-Klassifizierungsproblem.
In Anbetracht der Ergebnisse scheint die Computer Vision eine hervorragende Alternative zur morphologischen Klassifizierung von Arten zu sein, wenn das Modell mit einer großen Anzahl von Bildern guter Qualität trainiert werden kann und die auf dem Bild erscheinenden Arten von einem Spezialisten taxonomisch klassifiziert wurden.
Verbesserungswürdige Bereiche
Diese Ergebnisse können auf verschiedene Weise verbessert werden. Zunächst sollte das Modell mit einem angemessenen Datensatz trainiert werden, d. h. mit Bildern besserer Qualität und einer detaillierteren taxonomischen Klassifizierung. Auch eine Erhöhung der Anzahl der Bilder pro Label (z. B. von 1000 auf 2000) könnte dazu beitragen, morphologische Klassifizierungsfehler aufgrund subjektiver menschlicher Bewertung zu reduzieren.
Eine weitere Verbesserung wäre die Erhöhung der Anzahl der Gattungen. Derzeit haben wir das Modell mit 15 der 41 Gattungen trainiert, die ~1000 Bilder pro Label enthielten. Außerdem kann die Genauigkeit erhöht werden, wenn das gleiche Modell trainiert wird, um weitere Bilder von Pilzen auf Artniveau zu identifizieren.
Schließlich könnte die Kombination der Merkmalsextraktion unseres Modells mit einem klassischen Klassifizierungsmodell wie Random Forest oder SVM die Klassifizierung verbessern.
Referenzen
- Y. LeCun, L. Bottou, Y. Bengio, and P. Haffner. 1998. „Gradient-based learning applied to document recognition“.Proceedings of the IEEE.
- K. Simonyan, A. Zisserman. 2015. „Very Deep Convolutional Networks for Large-Scale Image Recognition“. Proceedings of the ICLR.
- Tan, Mingxing, and Quoc Le. 2019. „EfficientNet: Rethinking Model Scaling for Convolutional Neural Networks“. Proceedings of Machine Learning Research 97.
- F. Pedregosa, G. Varoquaux, A. Gramfort, V. Michel, B. Thirion, O. Grisel, M. Blondel, P. Prettenhofer, R. Weiss, V. Dubourg, J. Vanderplas, A. Passos, D. Cournapeau, M. Brucher, M. Perrot and E. Duchesnay. 2011. „Scikit-learn: Machine Learning in Python“. Journal of Machine Learning Research.