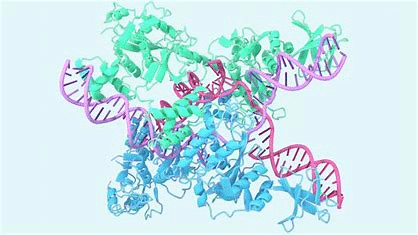
Proteine sind essenzielle Moleküle für das Leben und spielen eine entscheidende Rolle in vielen biologischen Prozessen. Sie sind in allen lebenden Zellen vorhanden und übernehmen eine Vielzahl lebenswichtiger Funktionen. Sie setzen sich aus Aminosäuren zusammen und falten sich in spezifische dreidimensionale Strukturen, die ihre Funktionen bestimmen.
Diese komplexen Strukturen erlauben den Proteinen, mit anderen Molekülen zu interagieren, chemische Reaktionen zu katalysieren, Zellsignale zu übertragen und den Zellen sowie Geweben strukturelle Unterstützung zu bieten.
Die genaue Struktur eines Proteins allein auf Grundlage seiner Aminosäuresequenz vorherzusagen, stellte lange eine große Herausforderung in der Biologie und Biochemie dar. Das Verstehen dieser Strukturen ist essentiell, da es ein vertieftes Verständnis des Wirkmechanismus der Proteine ermöglicht und die Entwicklung von Strategien zur Modulation ihrer Funktion fördert, was wiederum von entscheidender Bedeutung für die Entwicklung neuer Medikamente und Behandlungen ist.
In diesem Zusammenhang sticht AlphaFold, ein bahnbrechender Fortschritt im Bereich der Biologie, heraus.
Was ist AlphaFold?
AlphaFold ist ein KI-Programm (künstliche Intelligenz) von DeepMind, einer Tochtergesellschaft von Google, das sich auf Deep Learning spezialisiert hat. AlphaFold nutzt Neuronale Netzwerke, um die dreidimensionale Struktur von Proteinen basierend auf ihrer Aminosäuresequenz präzise vorherzusagen. Diese Innovation hat das Potential, unser Verständnis grundlegender biologischer Prozesse zu transformieren und einen beschleunigten Fortschritt in der Medizin und Biotechnologie zu fördern.
Die Herausforderungen der Proteinstrukturvorhersage
Die Vorhersage der Proteinstruktur stellt aufgrund mehrerer komplexer Faktoren eine erhebliche Herausforderung in der molekularen Biologie dar.
1. Vielfalt der Sequenzen und Strukturen:
Bis zum heutigen Tag sind mehr als 200 Millionen Proteine bekannt, und jährlich werden viele weitere entdeckt. Jedes dieser Proteine besitzt eine einzigartige dreidimensionale Form.
Proteine bestehen aus 20 verschiedenen Typen von Aminosäuren, die in Sequenzen angeordnet sind, welche in ihrer Länge und Zusammensetzung variieren. Diese Vielfalt führt zu einer enormen Anzahl possibler dreidimensionaler Strukturen, was die präzise Vorhersage dieser Strukturen extrem schwierig macht.
2. Grenzen der experimentellen Methoden:
Um die Struktur von Proteinen zu bestimmen, stehen verschiedene experimentelle Methoden zur Verfügung, wie die Röntgenkristallographie oder die Kernspinresonanz (NMR). Doch sind diese Methoden zeitaufwändig, kostenintensiv und nicht immer erfolgreich.
Es gibt Proteine, für die es schwierig oder sogar unmöglich ist, genaue strukturelle Daten durch traditionelle experimentelle Methoden zu gewinnen. Zu diesen Proteinen gehören solche, die sehr groß, flexibel sind oder sich schwer kristallisieren lassen.
Deshalb haben Wissenschaftler über Jahrzehnte hinweg nach einer zuverlässigen Methode gesucht, um die Struktur eines Proteins allein aus seiner Aminosäuresequenz zu determinieren.

Der Erfolg von AlphaFold
Im Wettkampf CASP (Critical Assessment of Structure Prediction), der alle zwei Jahre stattfindet, werden die Methoden zur Vorhersage dreidimensionaler Proteinstrukturen bewertet.
Zu diesem Zweck werden neu experimentell bestimmte Proteinstrukturen ausgewählt, die noch nicht veröffentlicht wurden, als Ziele. In den darauffolgenden Wochen müssen die verschiedenen teilnehmenden Teams die vorhergesagten Strukturen dieser Proteine mittels ihrer Methoden vorlegen. Dann werden die Vorhersagen mit den tatsächlichen experimentellen Strukturen verglichen, um die Genauigkeit der verschiedenen Vorhersagemethoden zu beurteilen.
2018 trat DeepMind unter den teilnehmenden Teams auf. Schon bei dieser Session (CASP13) zeigte sich AlphaFold allem Wettbewerb überlegen.
Bei CASP14 im Jahr 2020 übertraf AlphaFold alle anderen Teams mit einer bisher unerreichten Genauigkeit, die mit den traditionellen experimentellen Methoden vergleichbar war. Dieser Erfolg wurde als bedeutender Durchbruch in dem Bereich gefeiert.
Wie funktioniert AlphaFold?
AlphaFold kombiniert Deep-Learning-Technologien mit struktureller Modellierung, um die Proteinstrukturen vorherzusagen. Hier sind die Hauptphasen des Prozesses:
- Dateneingabe : Die lineare Sequenz von Aminosäuren des Zielproteins dient als Eingabe. AlphaFold erstellt multiple Sequenz-Alignments (MSA), um ähnliche Sequenzen in Protein-Datenbanken zu finden, was evolutionäre Informationen liefert.
- Modellierung : Mittels Deep-Learning-Modellen, inklusive Transformer, analysiert AlphaFold die Beziehungen zwischen den Aminosäuren. Transformer sind in der Lage, langreichweitige Beziehungen in Sequenzen zu handhaben, was unentbehrlich ist, um die Interaktionen zwischen in der linearen Sequenz weit entfernten, aber in der 3D-Struktur nah beieinander liegenden Resten vorherzusagen.
- Vorhersage von Abständen und Winkeln : AlphaFold sagt die Abstände zwischen den Aminosäurepaaren sowie die Winkel der chemischen Bindungen vorher, was entscheidend für die Bestimmung der dreidimensionalen Form des Proteins ist.
- Strukturelle Montage : Mit den Vorhersagen von Abständen und Winkeln montiert AlphaFold die dreidimensionale Struktur des Proteins, indem es eine Energie-Funktion minimiert, die unrealistische Konfigurationen sanktioniert.
- Bewertung der Vorhersage : Die vorhergesagte Struktur wird bezüglich ihrer Genauigkeit mit den vorhandenen experimentellen Daten bewertet und Verfeinerungstechniken werden eingesetzt, um die Qualität des Modells zu erhöhen.

Anwendungen von AlphaFold
Durch die schnelle und präzise Vorhersage der Proteinstrukturen ermöglicht AlphaFold neue Möglichkeiten für die biomedizinische Forschung und die pharmazeutische Entwicklung. Beispiele hierfür umfassen:
- Medikamentenentwicklung : Das Verständnis von Proteinstrukturen erleichtert das Design von Medikamenten, die gezielt bestimmte Proteine ansprechen, welche an Krankheiten beteiligt sind.
- Synthetische Biologie : Forscher können neue Proteine mit spezifischen Funktionen für industrielle oder umweltaffine Anwendungen erschaffen.
- Grundlagenforschung : Das Verstehen von Proteinstrukturen hilft dabei, fundamentale biologische Mechanismen zu erkennen und neue therapeutische Zielstrukturen zu identifizieren.
Teilung über die AlphaFold-Datenbank
AlphaFold verpflichtet sich, seine Technologie der Forschungsgemeinschaft zugänglich zu machen. Zu diesem Zweck hat DeepMind die AlphaFold Protein Structure Datenbank erstellt, die auf den Vorhesserungen von AlphaFold basiert.
Diese Datenbank ist kostenlos zugänglich und ermöglicht Forschern weltweit, auf diese Daten zuzugreifen und sie für ihre eigenen Forschungsarbeiten zu verwenden.
Die Datenbank umfasst mehr als 350.000 Strukturen, einschließlich 20.000 bekannter Proteine des menschlichen Körpers sowie Proteome anderer für die biologische Forschung relevanter Organismen, einschließlich Hefen und Mäusen.
Fazit
So verdeutlicht der Erfolg von AlphaFold bei der Vorhersage von Proteinstrukturen das revolutionäre Potential der künstlichen Intelligenz und des Deep Learnings in der wissenschaftlichen Forschung.
Um mehr über Deep-Learning-Technologien zu erfahren und eine Ausbildung in den Berufen der Datenwissenschaft zu beginnen, besucht DataScientest.