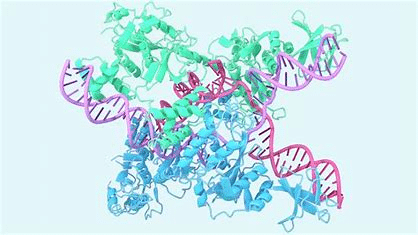
Les protéines sont des molécules essentielles à la vie, jouant un rôle crucial dans de nombreux processus biologiques. Elles sont présentes dans toutes les cellules vivantes et remplissent une multitude de fonctions vitales. Elles sont constituées d'acides aminés et se déploient dans des structures tridimensionnelles spécifiques qui déterminent leur fonction.
Ces structures complexes permettent aux protéines d’interagir avec d’autres molécules, de catalyser des réactions chimiques, de transmettre des signaux cellulaires et de fournir un soutien structurel aux cellules et aux tissus.
Cependant, prédire la structure exacte d’une protéine à partir de sa séquence d’acides aminés a longtemps été un défi majeur en biologie et en biochimie. La compréhension de cette structure est essentielle, car elle permet de mieux comprendre le mécanisme d’action des protéines et d’élaborer des stratégies pour moduler leur fonction, ce qui est crucial pour le développement de nouveaux médicaments et traitements.
C’est dans ce contexte que se distingue AlphaFold, une avancée révolutionnaire dans le domaine de la biologie.
Qu’est-ce que Alphafold ?
AlphaFold est un programme d’intelligence artificielle (IA) créé par DeepMind, une filiale de Google spécialisée dans l’apprentissage profond. AlphaFold utilise des réseaux de neurones pour prédire avec précision la structure tridimensionnelle des protéines à partir de leurs séquences d’acides aminés. Cette innovation a le potentiel de transformer notre compréhension des processus biologiques fondamentaux et d’accélérer les progrès en médecine et en biotechnologie.
Les défis de la prédiction de la structure des protéines
La prédiction de la structure des protéines représente un défi considérable en biologie moléculaire en raison de plusieurs facteurs complexes.
1. Diversité des séquences et structures :
À ce jour, plus de 200 millions de protéines sont connues, et de nombreuses autres sont découvertes chaque année. Chacune d’elles possède une forme tridimensionnelle unique.
En effet, les protéines sont constituées de 20 types d’acides aminés différents, arrangés en séquences pouvant varier en longueur et en composition. Cette diversité génère une multitude de structures tridimensionnelles possibles, rendant la prédiction précise de ces structures extrêmement difficile.
2. Limites des méthodes expérimentales :
Pour déterminer la structure des protéines, il existe différentes méthodes expérimentales telles que la cristallographie aux rayons X ou encore la résonance magnétique nucléaire (RMN). Ces méthodes sont néanmoins longues, coûteuses et ne réussissent pas toujours.
De plus, il existe des protéines pour lesquelles il est difficile, voire impossible, d’obtenir des données structurelles précises par des méthodes expérimentales traditionnelles. Ces protéines incluent celles qui sont très grandes, très flexibles, ou qui ne cristallisent pas facilement.
C’est pourquoi, pendant des décennies, les scientifiques ont tenté de trouver une méthode permettant de déterminer de manière fiable la structure d’une protéine à partir de sa seule séquence d’acides aminés.

Le succès de AlphaFold
La compétition CASP (Critical Assessment of Structure Prediction) est un évènement qui se tient tous les deux ans et qui vise à évaluer les méthodes de prédiction des structures tridimensionnelles des protéines.
Pour cela, des structures de protéines nouvellement déterminées expérimentalement (mais non encore publiées) sont sélectionnées comme cibles. Au cours des semaines suivantes, les différentes équipes participantes doivent fournir les structures de ces protéines prédites par leur méthode. Puis, les prédictions sont comparées aux structures expérimentales réelles, permettant d’évaluer la précision des différentes méthodes de prédiction.
En 2018, DeepMind apparaît parmi les équipes participantes. Dès cette session (CASP13), AlphaFold s’est avéré plus performant que tous ses concurrents.
Lors de CASP14, en 2020, AlphaFold a surpassé toutes les autres équipes avec une précision jamais vue auparavant, atteignant des niveaux comparables aux méthodes expérimentales traditionnelles. Ce succès a été salué comme une percée majeure dans le domaine.
Comment fonctionne AlphaFold ?
AlphaFold utilise une combinaison de techniques de deep learning et de modélisation structurelle pour prédire les structures protéiques. Voici les étapes principales du processus:
- Entrée des données : La séquence linéaire d’acides aminés de la protéine cible est fournie comme entrée. AlphaFold génère des alignements de séquences multiples (MSA) pour trouver des séquences similaires dans les bases de données de protéines, fournissant des informations évolutives.
- Modélisation : AlphaFold utilise des modèles de deep learning, y compris des transformateurs, pour analyser les relations entre les acides aminés. Les transformers sont capables de gérer les relations longues distances dans les séquences, ce qui est crucial pour prédire les interactions entre résidus éloignés dans la séquence linéaire mais proches dans la structure 3D.
- Prédiction de distances et d’angles : AlphaFold prédit les distances entre les paires d’acides aminés ainsi que les angles des liaisons chimiques, ce qui aide à déterminer la forme 3D de la protéine.
- Assemblage structurel : En utilisant les prédictions de distances et d’angles, AlphaFold assemble la structure tridimensionnelle de la protéine en minimisant une fonction d’énergie qui pénalise les configurations irréalistes.
- Évaluation de la prédiction : La structure prédite est évaluée pour sa précision par rapport aux données expérimentales disponibles, et des techniques de raffinement sont utilisées pour améliorer la qualité du modèle.

Applications de AlphaFold
En permettant une prédiction rapide et précise des structures protéiques, AlphaFold ouvre des perspectives nouvelles pour la recherche biomédicale et pharmaceutique. Par exemple :
- Développement de médicaments : La connaissance des structures protéiques facilite la conception de médicaments ciblant des protéines spécifiques impliquées dans des maladies.
- Biologie synthétique : Les scientifiques peuvent concevoir des protéines nouvelles avec des fonctions spécifiques pour des applications industrielles ou environnementales.
- Recherche fondamentale : Comprendre la structure des protéines aide à élucider les mécanismes biologiques sous-jacents et à découvrir de nouvelles cibles thérapeutiques.
Partage via la base de données AlphaFold
AlphaFold s’est engagé à partager leur technologie AlphaFold avec la communauté des chercheurs. Pour cela, DeepMind a mis en place la base de données AlphaFold Protein Structure faite à partir des prédictions de AlphaFold.
Cette base de données est disponible gratuitement, permettant ainsi aux chercheurs du monde entier de consulter et d’utiliser ces données pour leurs propres recherches.
Elle contient plus de 350 000 structures dont 20 000 protéines connues du corps humain mais aussi les protéomes d’autres organismes importants pour la recherche biologique, dont la levure ou encore la souris.
Conclusion
Ainsi, le succès d’AlphaFold dans la prédiction de la structure des protéines illustre le potentiel révolutionnaire de l’intelligence artificielle et du deep learning dans la recherche scientifique.
Pour en savoir plus sur les technologies de deep learning et vous former aux métiers de la Data, rejoignez DataScientest.